本年のノーベル賞(なかでも化学賞と物理学賞)は、恐らく多くの人にとって驚きを持って迎えられました。いずれの賞も、人工知能AI、特に機械学習とそれの応用についての業績に対する受賞であったからです。物理学賞は「人工ニューラルネットワークによる機械学習を可能にする基礎的な発見と発明」に対する受賞で、化学賞は「コンピュータによるタンパク質のデザインと構造予測」に対する受賞となりました[1]。ニューラルネットワークとは、人間の脳の神経細胞のネットワークを模倣したもので、特に多層のニューラルネットワークを利用するディープラーニング(深層学習)は、いまやコンピュータが様々なデータから学習することで問題に対処する「機械学習」の技術の中心となっています。物理学賞はこの機械学習の基礎研究に対しての業績が認められたものです。
化学賞の内容を説明するには、タンパク質の構造についておさらいする必要があります。タンパク質はアミノ酸が鎖状に結合してできた高分子です。20種類のアミノ酸が決まった順番につながっていき、体の中で様々な機能を司るタンパク質となります(図1)。しかし、タンパク質が酵素だったり、物質の運搬だったりの機能を発揮するには、単に鎖状に特定の順番につながる(これを一次構造といいます)だけではダメで、それぞれのタンパク質に固有な立体構造とならなければなりません。立体構造を解明するためには、X腺を使う方法などいくつかの方法がありますが、いずれも熟練した研究者が手間を掛けて行う必要があります。
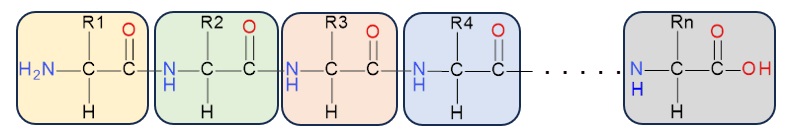 図1 アミノ酸が結合してできるタンパク質の一次構造。それぞれの色地が各アミノ酸部分を表し、R1~Rnにはそれぞれのアミノ酸に固有の基が入る。
図1 アミノ酸が結合してできるタンパク質の一次構造。それぞれの色地が各アミノ酸部分を表し、R1~Rnにはそれぞれのアミノ酸に固有の基が入る。
例えば最も古くその構造が解明されたタンパク質であるインスリンは、全部で51個のアミノ酸が結合した比較的小さなタンパク質(実際には21個のアミノ酸が結合した鎖と30個が結合した鎖の2本からなっています)ですが、図2や図3に示したような特別の立体構造となってはじめて血糖値の制御という機能を発揮するわけです。
図2 インスリンの立体構造。各球の色は以下の原子を表す。炭素(灰色)、赤(酸素)、青(窒素)、黄色(硫黄)、水素は省略してある。この図では硫黄原子間の結合によって2つの鎖がつながれていることが分かる。
データは https://www.rcsb.org/structure/3w7y から得た。
図3 インスリンの立体構造。図2と同じ構造をシンプルに表している。ピンクのリボンはアミノ酸がらせん状につながっている構造を表し、紫の矢印部分はアミノ酸が平面上につながっていることを表す。図2に示した硫黄原子間の結合は省いてある。
アミノ酸の配列の一次構造とタンパク質の立体構造の関係について解き明かすことは、極めて困難な課題でありました。しかし20世紀末になると、コンピュータを利用してこの問題を解く試みが始まり、比較的短いタンパク質については構造の予測もできるようになってきました。そして2003年に、今回のノーベル化学賞受賞者の一人であるDavid Baker博士(1962-、ワシントン大学教授)は、画期的な研究成果[2]を報告しました。彼らはこれまでのタンパク質に類似性のない構造を持つ93個のアミノ酸が結合したタンパク質をデザインし、彼らが開発したRosettaというソフトウエアでその構造を予測しました。また実際にそのタンパク質を合成してX線構造解析の手法で構造を決定し、計算による予測とよく類似していることを示したのです。
さらにBaker博士らは、天然にない酵素のデザインを行い、実際にそれが機能することを実証したり、特定の分子と強固に結合するタンパク質を新たにデザインしたりするような研究も行い、コンピュータを利用した、新たなタンパク質のデザインが広範囲の応用に結びつくことを示しました。
また、タンパク質の一次構造から立体構造を導き出す研究も20世紀末から少しずつ進んできましたが、大きなサイズのタンパク質への適用は不可能と思われてきました。しかしここ数十年の間に実験的に立体構造が解明されたタンパク質の数が増え、AIをこの分野に適用しようという試みがなされるようになってきました。そしてニューラルネットワークを利用した機械学習が2016年頃から取り入れられるようになり、この分野は急速に進歩していくことになります。そのような中、英国のDeepMind社(この企業はチェスや囲碁を行わせるプログラミングで有名でした。現在の名称はGoogle DeepMind社)のDemis Hassabis博士(1976-)が率いるチームが、2018年に深層学習の手法を用いてAlphaFold(AF)というソフトウエアを発表しました。AFはタンパク質の構造をこれまでにない精度で予測することができました。2020年にはもう一人の受賞者であるJohn Jumper博士(1985-)も加わったチームから、AlphaFold2(AF2)が発表され、実験的に実際のタンパク質の構造を決定するときの精度に匹敵する精度で構造を予測することができるようになり、研究者を驚かせました[3]。このAF2ソフトウエアは、論文においてそのプログラム本体(ソースコード)が完全に公開され、このことによってこの研究に大きな信頼を与えたのです。先のBaker博士らもAF2の考え方を取り入れた新たなソフトウエアを発表しています。
2024年にはAF3が発表され、さらに計算速度とその適用範囲が広がりました。このソフトウエアのソースコードは公開されておらず、その点で批判もあるようですが、非商用目的であれば誰でもその威力を試すことができます[4]。
AIの研究分野への進展は恐るべきものです。誰がこのような分野でノーベル化学賞が授与されると予想したでしょうか。恥ずかしながら私はまだまだAIは発展途上であり、そのお世話になることは当分ないと思っていました。なにせ自分の名前をChatGPTに入れるととんでもない答えが返ってくるものですから。しかし我々はAIを上手く使っていくことをすぐにでももっと真剣に考える時代となったようです。それではまた次回。
[1] ノーベル化学賞の内容はノーベル財団のサイト、https://www.nobelprize.org/prizes/chemistry/ に詳細が掲載されています。そのサイト中に“Scientific background to the Nobel Prize in Chemistry 2024“ という記事があり、今回の原稿の内容はほとんどこの文章によっています(2024年10月25日閲覧)。
[2] B. Kuhlman, G. Dantas, G. C. Ireton, G. Varani, B. L. Stoddard, D. Baker, Science 2003, 302, 1364-1368.
[3] J. Jumper, R. Evans, A. Pritzel, T. Green, M. Figurnov, O. Ronneberger, K. Tunyasuvunakool, R. Bates, A. Žídek, A. Potapenko, A. Bridgland, C. Meyer, S. A. A. Kohl, A. J. Ballard, A. Cowie, B. Romera-Paredes, S. Nikolov, R. Jain, J. Adler, T. Back, S. Petersen, D. Reiman, E. Clancy, M. Zielinski, M. Steinegger, M. Pacholska, T. Berghammer, S. Bodenstein, D. Silver, O. Vinyals, A. W. Senior, K. Kavukcuoglu, P. Kohli and D. Hassabis, Nature, 2021, 596, 583–589.
[4] https://alphafoldserver.com/about ここで登録することで、非営利目的ならAF3を使うことができます。実際筆者もインスリンのアミノ酸配列を入力してその立体構造が得られることを確認しました。
本稿では当初、このソフトウエアのソースコードは公開されていないように書きましたが、読者の方から公開されたとのご指摘をいただき、その部分の表現は削除いたしました。ご指摘に感謝いたします。
改訂:2024年12月11日
AF3に関する記載について、修正(打消し線の追加)および注釈[4]への追記を行いました。
坪村太郎
最新記事 by 坪村太郎 (全て見る)
- 2025年 ノーベル化学賞:3次元空間を自在にデザイン。空孔をもつ錯体化合物 - 2025年12月3日
- 火星で生命の痕跡か - 2025年11月3日
- 宇宙で作った味噌 - 2025年10月6日